En numerosas ocasiones, durante las conversaciones sobre genética con ganaderos, colegas y estudiantes acerca del interesante tema de la selección de los toros que harán parte del programa de mejoramiento genético en una ganadería surgen interrogantes básicos acerca de la terminología utilizada en los catálogos de inseminación artificial y páginas relacionadas; por lo cual es importante conocerlos y entender claramente cuál es la información que nos suministra cada uno de ellos y cómo la podemos aplicar en los programas de apareamiento dirigido a nivel de finca
La genética es la rama de la biología que se encarga de estudiar las características o rasgos que se transmiten de padres a hijos, por lo que se conocen como hereditarios. Al interior de cada una de las células de un organismo, el contenido nuclear contiene unas estructuras denominadas cromosomas, los cuales están compuestos por secuencias muy largas de cadenas de pares de bases (adenina – citosina – guanina – timina) enlazadas con un azúcar en el centro (desoxirribosa) conocidas como el ADN (ácido desoxi ribonucleico) conformado por millones de pares de bases ordenadas a manera de escalera de caracol que interactúan para mantener su arquitectura; existen unas secuencias de pares de bases o unidades moleculares de la herencia genética que se conocen como los genes, encargados de codificar cada uno de los rasgos de un individuo. Cada uno de los genes están ubicados dentro de los cromosomas en unas posiciones determinadas y específicas que se conocen como “locus” y el estudio de todas estas estructuras es lo que se conoce como genómica.
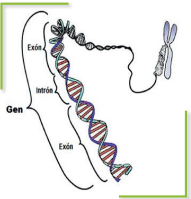
Desde hace muchos años la ciencia ha mostrado interés particular en “desenrollar” esas cadenas de pares de bases para establecer la secuencia completa de las mismas en cada especie, lo que se conoce como el “genoma” o el mapa genómico del individuo y así saber en qué posición se encuentran los segmentos que codifican cada característica.
Haplotipos, genes recesivos y principales códigos genéticos
A continuación, se presenta una traducción y adaptación de un artículo encontrado en la página del NBDC (National Bovine Data Centre, del Reino Unido), que los lectores pueden consultar en su versión original en: www:ndbc.uk Es una página diseñada para ayudar a explicar los códigos genéticos actuales que aparecen después de los nombres de los animales de razas lecheras registrados en pedigríes, hojas informativas web, catálogos de IA, boletines genéticos y listas principales.
Las siguientes figuras pueden ayudar a visualizar la localización de estos componentes genéticos; seguidas por algunas definiciones de los términos más comunes que se utilizarán en la descripción y explicación de los códigos genéticos de los bovinos que se mencionarán más adelante.
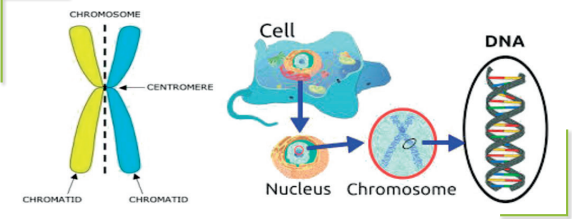
Cromosoma: Es una gran unidad de ADN que contiene muchos genes y cada cromosoma está formado por un par de cromátides. Los bovinos presentan un número de 60 cromosomas en total (2n=60) que se encuentran en la mayoría de las células del cuerpo, a excepción de los espermatozoides y óvulos, en las cuales las cromátidas existen como entidades únicas no apareadas (n=30). Por lo tanto, un animal hereda la mitad de cada cromosoma y, por lo tanto, una copia de cada gen de cada padre.
Gen: Es una secuencia de ADN que codifica (es decir, es el manual de instrucciones) para un aminoácido en particular; los aminoácidos son los componentes básicos de las proteínas. Cada animal tiene dos copias de cada gen (que pueden variar en su presentación).
Alelo: Es una forma o versión de un gen. Un animal puede tener el mismo alelo en ambos cromosomas (homocigotos) o diferentes alelos (heterocigotos). Los genes pueden tener múltiples alelos, como el gen MCR1 responsable de algunas de las variaciones de color del pelaje rojo, sin embargo, el 2 es el más común. Un animal solo puede heredar un alelo de cada padre.
Alelo recesivo: Es aquel que solo tiene efecto cuando un animal hereda esta versión del gen de cada padre. Un alelo dominante afecta a un animal cuando se heredan una o dos versiones. La mayoría de los alelos dañinos son recesivos.
SNP (Single Nucleotide Polymorphism o Polimorfismo de Nucleótido Simple): es una unidad única de ADN que varía entre animales dentro de la misma raza o especie. Un SNP puede estar ubicado dentro de un gen o en una región entre genes en un cromosoma. Los SNP son los marcadores utilizados para genotipificar animales en todos los programas establecidos para la selección genómica.
Haplotipo: Es un grupo de SNP o alelos ubicados cerca uno del otro en el cromosoma, que generalmente se heredan juntos. A través de los años la ciencia ha logrado encontrar y demostrar la presencia de varios haplotipos que pueden llegar a afectar significativamente la producción y/o reproducción en las principales razas de lechería especializada, diversas investigaciones han reportado la presencia de haplotipos asociados con disminución en las tasas de desempeño reproductivo, principalmente en lo que tiene que ver con mortalidad embrionaria y abortos tempranos.
Hasta el momento se conocen seis haplotipos defectuosos, identificados mediante pruebas genómicas, con impacto negativo en la fertilidad en la raza Holstein, que son HH1, HH2, HH3, HH4, HH5 y HH6. De manera similar, el haplotipo JH1 se informa en la raza Jersey, así como BH2 en Brown Swiss y AH1 y AH2 en Ayrshire.
Se desconocen las razones exactas por las que los haplotipos afectan la fertilidad; sin embargo, se cree que la herencia del mismo haplotipo defectuoso de cada padre da como resultado una concepción fallida o una muerte embrionaria temprana.
Los animales que portan una versión de un haplotipo defectuoso se codifican con una C (Carrier). Por ejemplo, un animal que porta HH1 se codifica como HH1C. Los animales identificados como libres del haplotipo se codifican con una T, por ejemplo: HH1T, que en teoría debería ser el código que se busca al seleccionar un toro.
Heredabilidad de un haplotipo
1. Si ambos padres son portadores de un haplotipo indeseable (HH1C) hay un 25% de probabilidad de que haya una descendencia afectada que no sobreviva al nacimiento; y de su descendencia viva, un tercio serán no portadores no afectados y dos tercios serán portadores, por ejemplo: Vaca HH1C (portadora = Rr) x Vaca HH1C (portadora = Rr)
R = Haplotipo normal / r = Haplotipo HH1 (que contiene la mutación causante)
2. Si se desconoce la condición genética madre, pero el abuelo y el toro son portadores no afectados de un haplotipo indeseable (HH1C): existe un 12,5% de probabilidad de que el embrión resultante no sobreviva hasta el nacimiento.
3. Si la vaca y el toro fueran portadores de diferentes haplotipos, por ejemplo. si la vaca fuera HH1C y el toro fuera HH2C, se podría esperar la siguiente descendencia resultante: - 25% no portadores de ambos (HH1T y HH2T))
- 25% portadores de uno (HH1C)
- 25% portadores del otro (HH2C)
- 25% portadores de ambos (HH1C y HH2C)
El mejor consejo es evitar el uso de toros que porten alguno de estos haplotipos defectuosos, ya que el uso de tales toros reducirá las tasas de concepción en el rebaño promedio. Y en un futuro cercano, cuando tengamos la posibilidad de analizar las hembras del hato, por medio de las pruebas genómicas, entonces tendremos la posibilidad de tomar decisiones más precisas con respecto al uso de un toro portador si la hembra no es portadora.



Humberto Guáqueta Munar. M.V. PhD. Ciencias & Salud Animal.
Universidad Nacional de Colombia












