La ancestría racial de un animal de origen multirracial nos indica aproximadamente qué parte de su genoma proviene de una raza u otra. Ejemplo de esto son las razas “sintéticas” que han sido producto reciente de cruzamientos dirigidos entre un par o más razas “puras”, con el objetivo de maximizar las propiedades zootécnicas de dichas razas parentales hacia una población lo más homogénea posible que manifieste esas ventajas productivas, reproductivas y/o de calidad.
Entre las razas sintéticas encontramos al Brangus, Simbrah, Girolando, entre otras. Además, tenemos individuos producto de cruzamientos dirigidos de razas puras hacia proporciones establecidas (1/4, 3/8, 5/8…) que tienden a manifestar ciertas características de una raza o de la otra, según el objetivo productivo.
El grado de ancestría racial de un animal respecto a sus razas parentales se ha estimado usualmente con el pedigrí del animal y de los cruzamientos que dieron su origen. Para ello, se infiere la proporción de “sangre” heredada en cada cruzamiento, conociendo la genealogía de los parentales y el orden en los cruzamientos realizados previamente para llegar a ese individuo. Como ejemplo, el cruzamiento de dos parentales “puros” de razas distintas, Charolais y Senepol producen una descendencia F1, 50% (1/2) de ancestría Charolais y 50% (1/2) Senepol. Para la segunda generación, al cruzar uno de esos F1 con un animal “puro” Senepol, la estimación nos indica que esos ejemplares F2, tendrían una ancestría 75% (3/4) Senepol y 25% (1/4) Charolais. Esta proporción de ancestría estimada es un promedio esperado de todas las crías producto de ese cruzamiento, no a nivel individual. Y a la final, esta estimación parte de varios supuestos, que no se cumplen o se malinterpretan después de la F1.
Uno de estos supuestos es que cada uno de los animales productos de cruzamientos de 2 o más razas “puras” o de F2 en adelante, tienen cada uno un porcentaje idéntico de ancestría racial de las razas, lo cual es incorrecto. A partir de la información genética y genómica de las últimas décadas hay evidencia que cada animal producto de ejemplares cruzados tiene un porcentaje diferente de ancestría racial de las razas que los originaron, como en bovinos (Jaafar et al. 2021) y en otras especies como caninos (Dreger et al. 2016). Como argumento, si analizamos con análisis genéticos/genómicos a un grupo de animales catalogados por un criador como 5/8-3/8 Brangus, todos tendrían una proporción diferente de ancestría racial Angus y Brahman. Es decir, en realidad ese concepto de 5/8-3/8 es una aproximación cruda de lo que en realidad tienen en su ADN dichos animales. Esto se debe a que hay procesos biológicos que recombinan la información genética en la meiosis y por ello no todos los hermanos completos son iguales en su genotipo y por lo mismo no todos tienen la misma proporción de genes de una u otra raza.

La recombinación genética
La recombinación genética es la clave para entender por qué cada ejemplar tiene información genética única, así sean hermanos completos y del porque la estimación racial por pedigrí está sesgada. Cuando se produce un gameto, ya sea espermatozoide u óvulo, las células germinales pasan por una serie de procesos biológicos para dividir la información genética a la mitad y que cada gameto tenga una combinación única de genes de los padres, lo que se conoce como meiosis y recombinación genética. En el proceso de división celular, el material genético de padre y madre se intercambia en diferentes puntos de los cromosomas (unidad empaquetada de ADN, los bovinos tienen 30 pares, uno de la madre y otro del padre) y luego de ese intercambio, se divide en cada gameto (óvulo o espermatozoide) con una combinación genética única para cada uno. Por esto en gran medida, es que todos los organismos diploides (2n, es decir con dos cargas genéticas una de padre y otra de madre) somos únicos en genética y en biología.
Por lo anterior, si usamos la información genética del animal tenemos un valor más “real” de ese componente racial de un individuo. Para esto, se requiere tener información genética de referencia de las poblaciones “puras” y del ejemplar que se requiere analizar. Para ello, se usan análisis de marcadores genéticos, es decir, partes en el ADN de genoma del animal que varían de un individuo a otro en una población determinada. Entre más marcadores genéticos se usen y mayor la cantidad de animales en la población de referencia, más exacta será la estimación de ancestría. Actualmente, es posible utilizar marcadores genéticos tipo microsatélites o tipo SNPs en chips de ADN para abarcar de unos miles a cientos de miles de marcadores.

Relación entre ancestría y ganado de leche
La relación entre la ancestría de animales cruzados y parámetros productivos en ganadería de leche se evidencian en trabajos como el grupo del Dr. Huson de la Universidad de Cornell (“Impact of genomic breed composition on production traits in crossbred dairy cattle” Jaafar et al. 2021). En este trabajo analizaron dos poblaciones de animales cruzados, una llamada ProCROSS (378 animales) de origen de razas Holstein (HOL), Montbelliard (MON) y Rojo Sueco (VKR) y otra llamada “Grazecross” (229 animales) cuyo origen se basó en Jersey (JER), Normando (NOR) y Rojo Sueco. Midieron en esos ejemplares 4 características: Producción de leche (MY), de grasa (FY), proteína (PY) y conteo de células somáticas (SCS) y genotiparon cada animal con un chip de 50K. La estimación de ancestría de cada ejemplar se ve en la figura 1. Cada ejemplar tiene un componente particular en su genoma de una u otra raza.
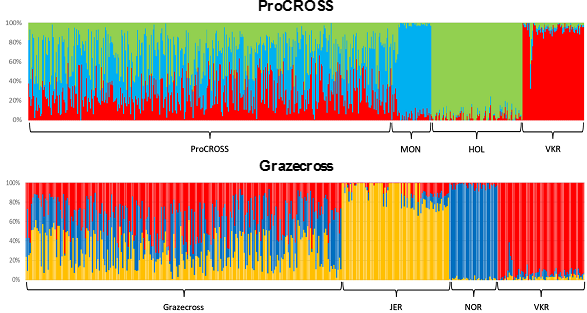
Figura 1. Análisis de ADMIXTURE que refleja la ancestría global de las razas para los individuos mezclados por ProCROSS y Grazecross y todas las poblaciones parentales para K = 3 (número de razas parentales definidas). Cada línea representa el componente genético de un animal. Tomado de Jaafar et al. 2021.
La comparación de la media de la composición global de la ancestría entre los animales de alto y bajo rendimiento mostró que la composición HOL desempeña un papel significativo en el MY y el FY en las poblaciones ProCROSS, mientras que en la población Grazecross, la composición VKR y NOR desempeñan un papel significativo en el rendimiento SCS (valor p < 0.05). Este hallazgo proporcionó la base para comprender el impacto de la ancestría específica de la raza en el rendimiento lechero del ganado cruzado.
Finalmente, estos hallazgos posiblemente se podrán aplicar también en ganadería de carne. Por lo cual, establecer los porcentajes de ancestría podrá ser un criterio de selección para mejorar las producciones de las unidades productivas en un futuro cercano.
Miguel Novoa-Bravo PhD.
Director Científico, Genética Animal de Colombia SAS.
miguelnovoa@geneticaanimal.co









